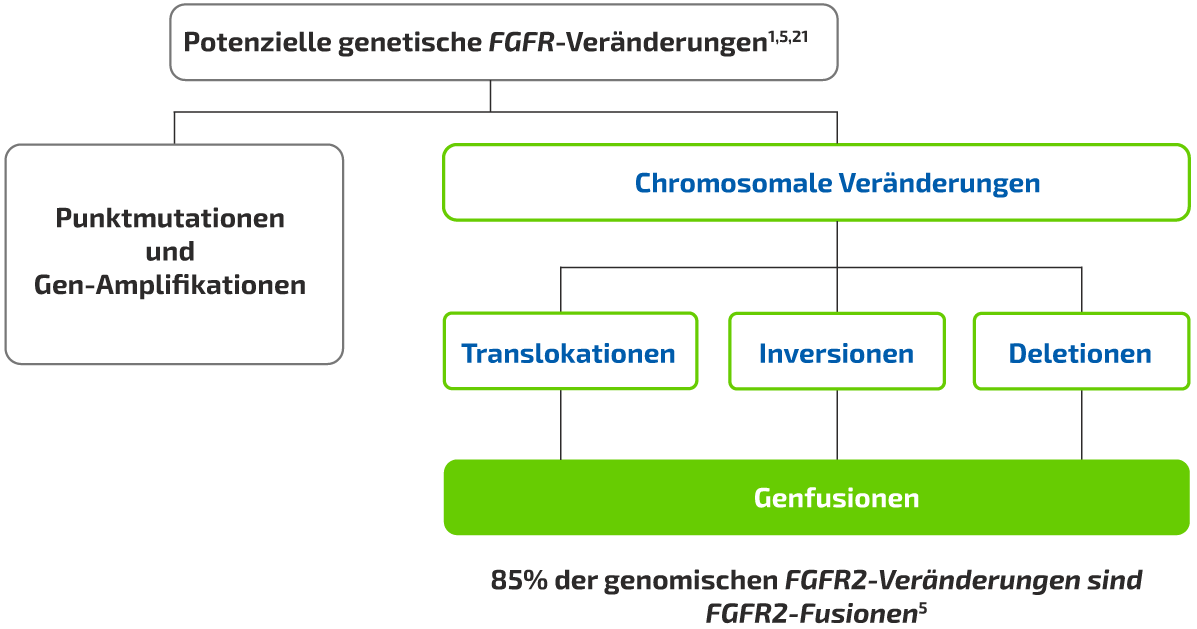
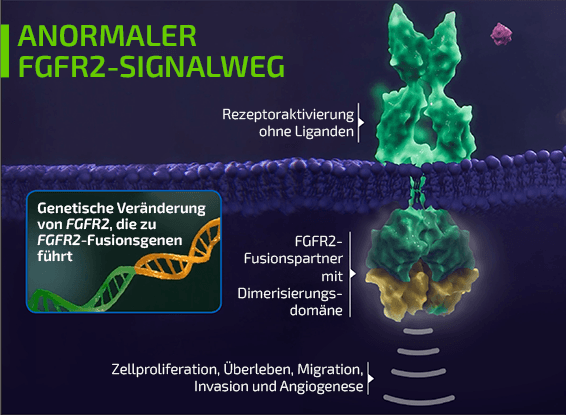
Genomische Veränderungen beim intrahepatischem Cholangiokarzinom (iCCA)
Genomische Veränderungen mit potenziellen therapeutischen Auswirkungen wurden bei ~40 % der Patienten mit iCCA identifiziert, was ein individuelleres Bild von der Biologie der Krankheit eines jeden Patienten ermöglicht.1
- Zu den zwei häufigsten genomischen Veränderungen im iCCA gehören Isocitrat-Dehydrogenase 1 (IDH1)-Mutationen und Fibroblasten-Wachstumsfaktor 2 (FGFR2)-Fusionen1,2
Beim iCCA sind zahlreiche potentiell zielgerichtet therapierbare, genomische Veränderungen vorhanden1*
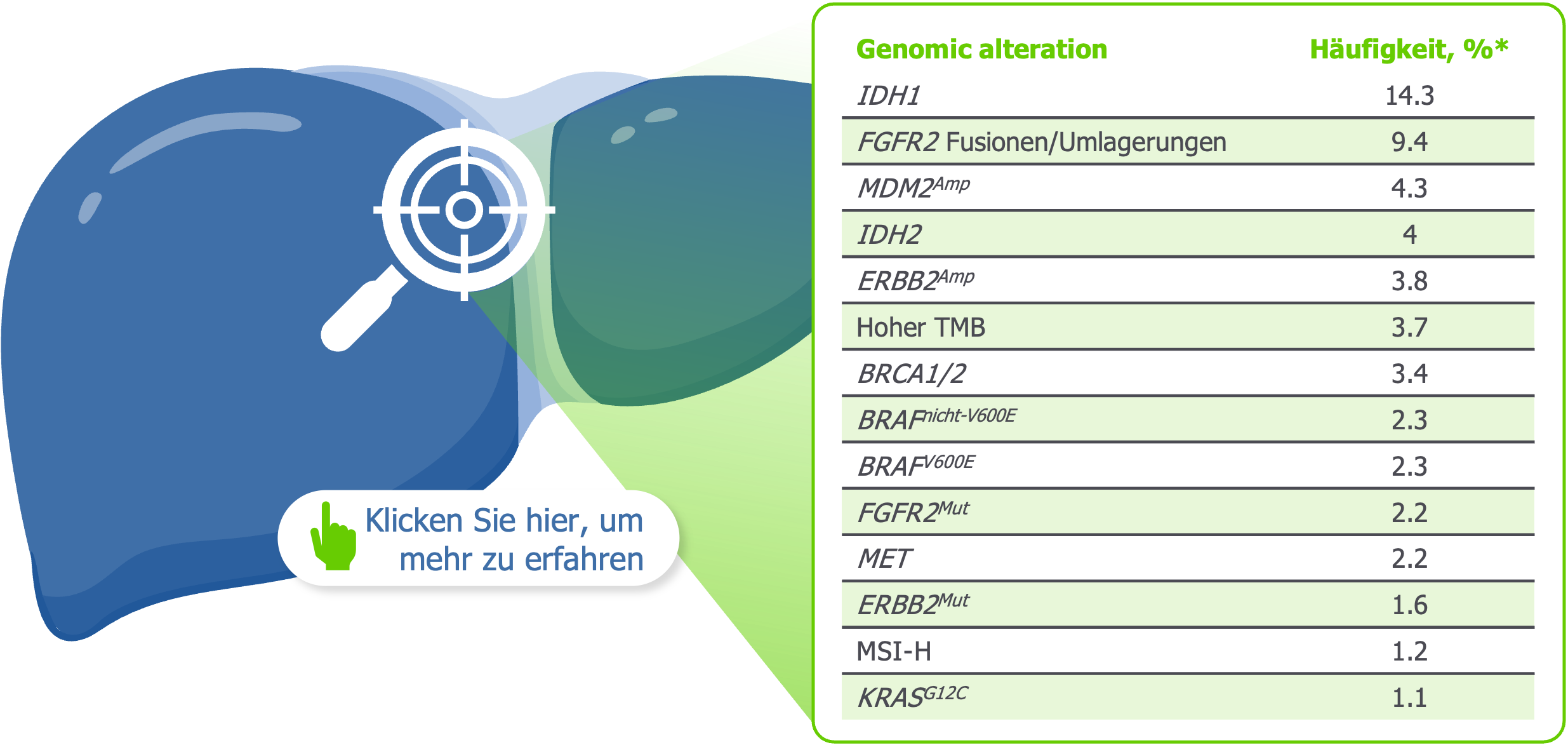
*Basierend auf einer retrospektiven Analyse von 6.130 Patienten mit iCCA aus der FoundationCORE-Datenbank, die eine diagnostische Panel-Sequenzierung auf der FoundationOne-Plattform erhielten. Kurze Varianten, Fusionen/Umlagerungen und Kopienzahlveränderungen in über 300 tumorassoziierten Genen wurden bewertet, und der TMB- und MSI-Status war für die Mehrheit der Kohorte verfügbar.1
amp, Amplifikation; BRAF, B-Raf-Protoonkogen; BRCA1/2, Brustkrebsgen 1/2; ERBB2, erb-b2-Rezeptor-Tyrosinkinase 2; ERK, extrazellulär signal-regulierte Kinase; FGFR2, Fibroblasten-Wachstumsfaktor-Rezeptor 2; iCCA, intrahepatisches Cholangiokarzinom; IDH1/2, Isocitrat-Dehydrogenase 1/2; KRAS, virales Onkogen-Homolog des Kirsten-Ratten-Sarkoms; MAPK, mitogen-aktivierte Proteinkinase; MDM2, Maus-Doppelminute-2-Homolog 2; MET, mesenchymales epitheliales Übergangsproto-Onkogen; MSI, Mikrosatelliteninstabilität; MSI-H, hohe Mikrosatelliteninstabilität; mut, Mutation; NADPH, Nicotinamidadenindinukleotidphosphat (reduziert); TMB, Tumormutationslast.
Abbildung übernommen aus Kendre G, et al. J Hepatol. 2023;78:614–26.1